基因融合(Gene Fusion )是指两个不同基因的部分序列或全部序列融合到一起,形成了一个新的基因。基因融合的常见机制有染色体易位、插入缺失及倒位等。越来越多的研究表明,基因融合与各种疾病,特别是癌症的发生发展紧密相关,甚至是一些癌症的直接诱因。因此对融合基因的精准检测,对于肿瘤患者的诊断、用药及预后等临床诊疗过程具有重要意义。
目前常用的融合检测手段有免疫组织化学( Immunohistochemistry,IHC) ,荧光原位杂交(Fluorescence In Situ Hybridization ,FISH),实时荧光定量PCR( Quantitative Real-time PCR ,qPCR) 及下一代测序技术( “Next-Generation" Sequencing technology,NGS )(图1 ),其中NGS 融合检测依据采用的检测底物又可分为DNA-NGS 及RNA-NGS ,不同的融合检测方法各有利弊(表1 )。
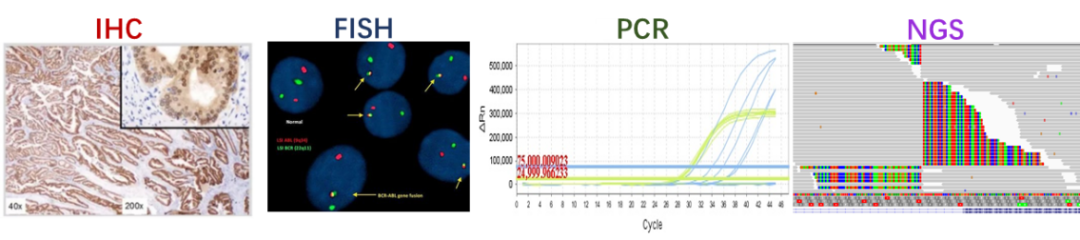
图1 常见的几种融合检测方法
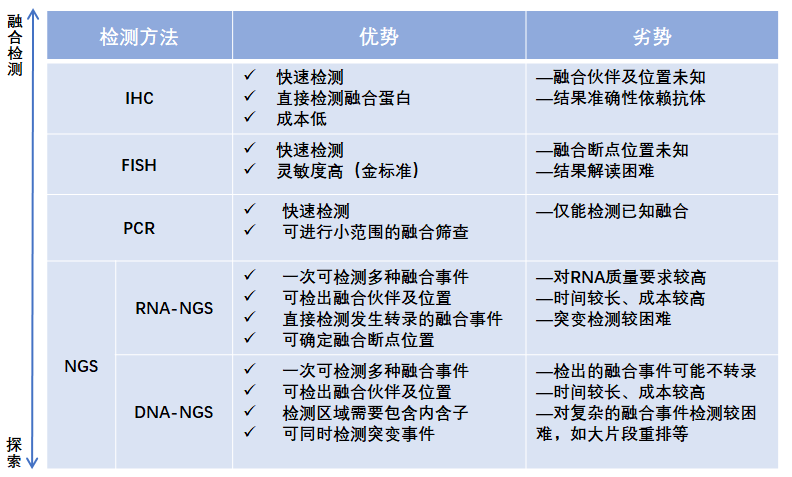
表1 常见的几种融合检测方法优劣势比较
以费城染色体样急性淋巴细胞白血病(ph-like ALL )为例,目前已经发现了近百种融合(图2) [1,2] 。传统的检测手段IHC 、FISH 及qPCR 受通量的限制,难以一次性对这么多种融合进行筛查,而NGS 恰恰具有这种优势,可一次性对多种融合进行大规模筛查。那么选择哪一种NGS 路线进行融合检测呢?
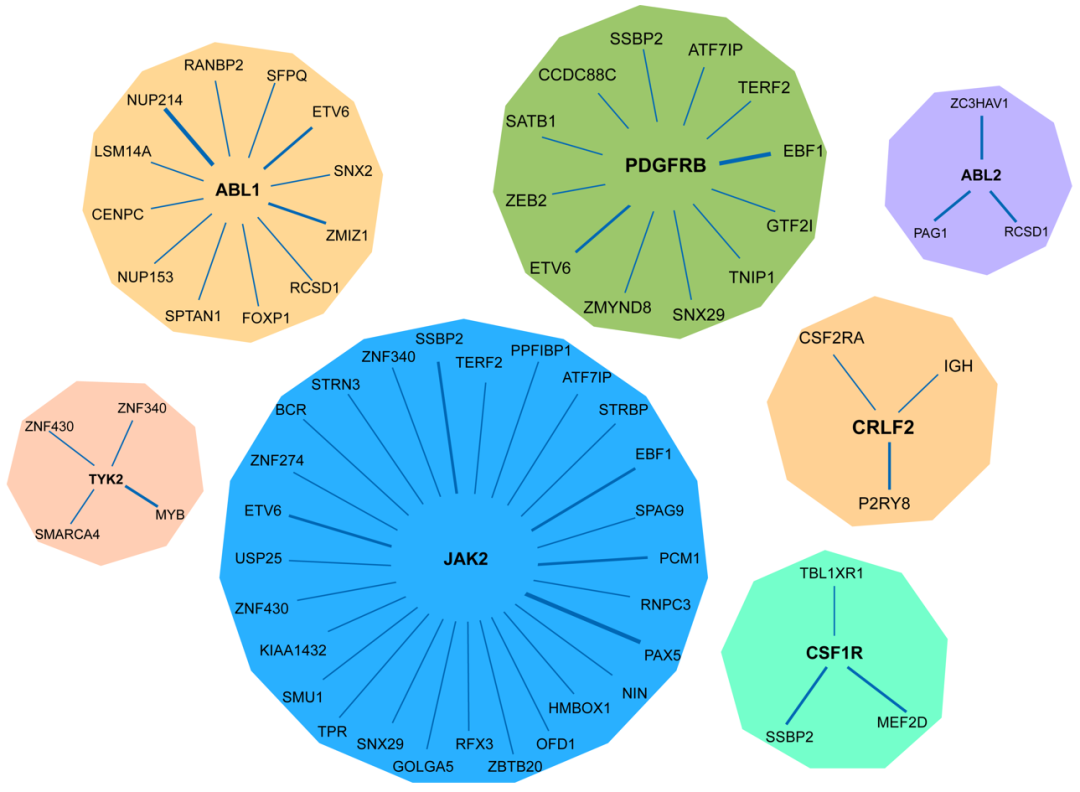
图2 Ph-like ALL主要的融合基因家族
在DNA 水平上,融合断点位置通常发生在较长的内含子区域,且融合的断裂点不同患者可能不同,因此采用DNA-NGS技术设计探针去抓取断裂点不可避免地存在一个问题,即:为保证准确地找到融合断裂点,探针设计需要覆盖非常冗长且含有大量重复序列的内含子区域,造成检测成本增加的同时,这些内含子区域可能存在高GC 、位于repeat区等一系列影响探针捕获效率及序列比对准确性问题 。 而相比DNA ,RNA 水平上融合基因表现为前后两个基因外显子之间的衔接,融合点相对固定(图3 )。这一特征使得精准设计探针更为容易 ,同时探针的设计更多位于外显子区,也极大避免了高GC 、重复区等对检测准确性的影响。
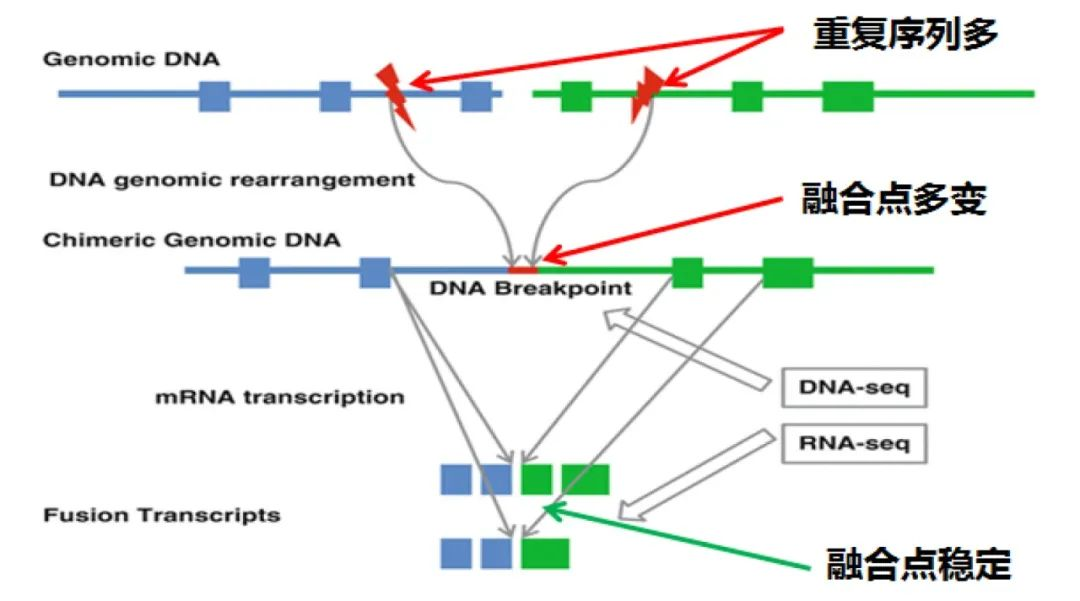
图3 融合基因示意图
另一方面,融合基因可能由于融合而导致表达激活,从而产生大量的RNA 融合转录本。典型的研究包括Seo等人发表的一项关于肺腺癌的转录特征和突变特征的研究 [3] ,该研究显示不管是ALK 融合、RET融合还是ROS1 融合,融合基因的RNA 表达量都显著上调(图4 )。其原因是融合基因保留了其激酶催化结构域的3’ 端和融合伴侣启动子的5’ 端,融合伴侣在自身发生转录的同时,其下游激酶区域同时发生了转录。即在生理状态下原来不应该转录的激酶区域,在形成融合基因后,其激酶区域转录被激活产生了大量的mRNA 。因此,在RNA 水平上检测融合基因比DNA 水平更敏感,基于RNA 进行融合检测的方法也越来越受到青睐。
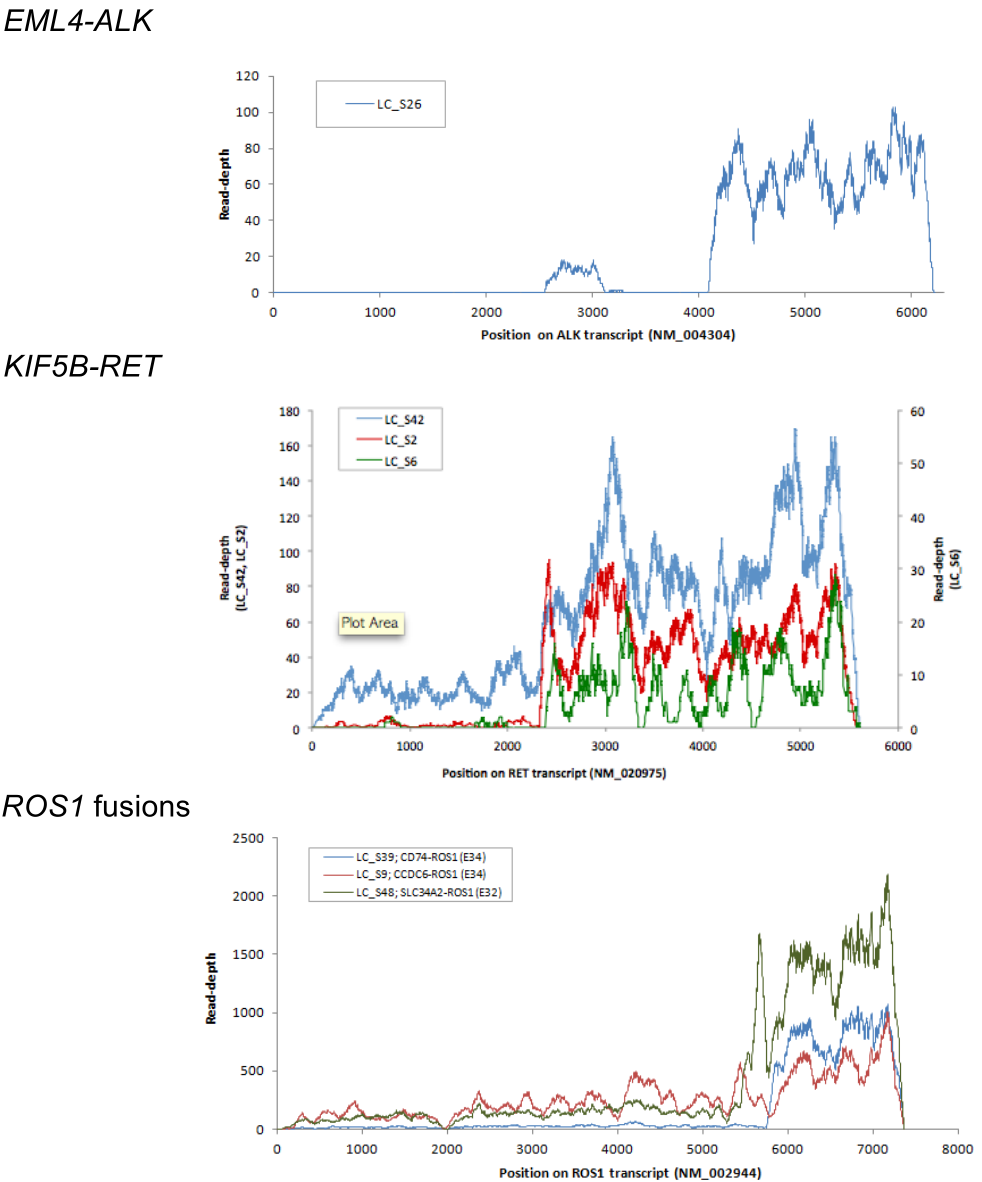
图4 融合转录本由于融合的发生表达量显著上调 [3]
具体到RNA-NGS ,主流的融合检测路线为RNA-seq,即通过高通量测序技术进行的转录组测序分析技术。常规RNA-seq 依据对RNA 的富集方式可分为polyA RNA-seq 及rRNA depletion RNA-seq。二者的区别在于前者以包被有oligodT 的磁珠将总RNA 中带有polyA 尾的mRNA 进行富集后构建文库,而后者采用rRNA 探针将总RNA 中的rRNA 进行富集后丢弃,对剩下的RNA 种类进行文库构建。由此可知,polyA RNA-seq 依赖RNA 的polyA 尾,要求RNA的完整度较高,对于容易发生降解的RNA 类型,如FFPE RNA 则效果不佳,而rRNA depletion RNA-seq则可以有效克服这一缺陷。然而,两种方法的共同问题在于,尽管都通过对数量占绝大多数(~90% )的rRNA 进行了去除(rRNA 不具有polyA 尾),即对携带有效信息的RNA 进行了一定程度的富集,但真核细胞的表达谱具有转录本数量众多且表达量高低差异大的特点。RNA-seq 测序结果分散在整个基因组中,不同基因测序深度差异显著,对转录水平偏低的转录本往往测序深度不足,相应的融合检测效果不佳 。 因此,在RNA-seq 基础上,衍伸出了靶向RNA-seq (Targeted RNA-seq ),又叫RNA 捕获测序(CaptureSeq ),是指通过寡核苷酸探针杂交,先对RNA文库进行序列靶向捕获,以富集感兴趣的转录本,再进行测序,从而实现只检测感兴趣的靶标基因RNA 转录本的方法(图5 )。这一方法可以提高目标转录本的测序深度,进行灵敏的基因发现、有效的转录本组装和准确的基因表达定量。相比标准RNA-seq ,具有高灵敏度、宽动态范围、低成本与高通量等优势。
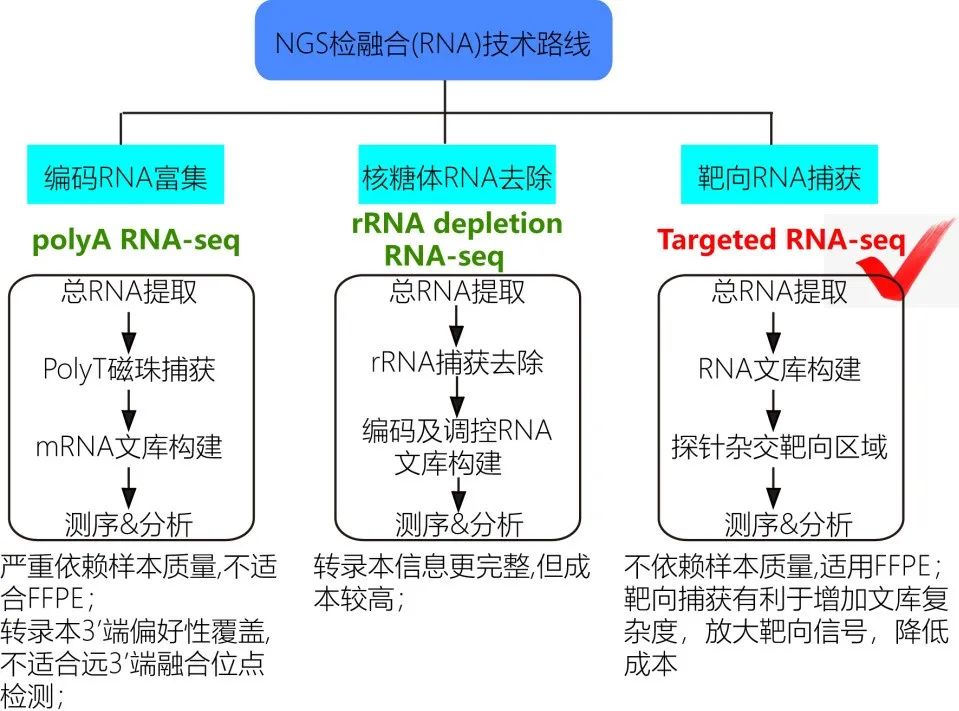
图5 三种RNA-NGS检融合技术路线
常规的靶向RNA-seq 是在标准RNA-seq的基础上增加探针杂交富集的过程(图6 ) [4,5] 。其中探针设计有两种形式,一种是基于外显子序列设计,另一种是基于转录本序列设计,两种设计形式的区别在于对外显子接合区(junction site )的捕获能力有一定差异。也可将两种形式结合起来,在基于外显子设计探针的基础上,增加基于对特定的融合转录本进行针对性的探针设计,直接捕获融合转录本的接合区,从而进一步提高融合检测灵敏度。
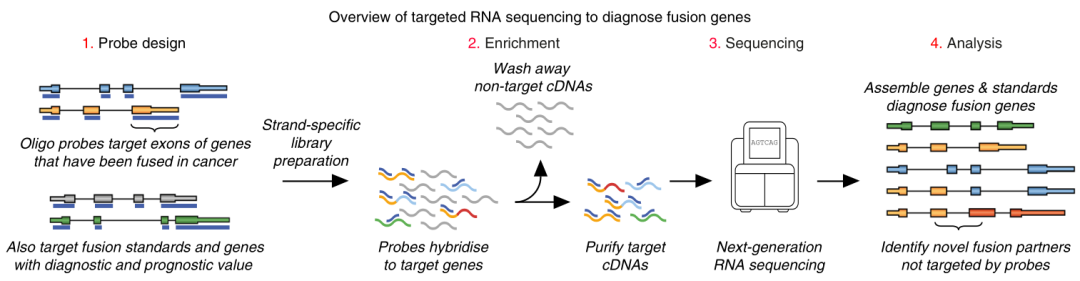
图6 靶向RNA-seq检测流程图 [4]
迈杰转化医学基于靶向RNA-seq 技术开发的血液肿瘤156基因融合检测产品HEMECDx panel 旨在检测156个血液肿瘤基因相关的融合,最低可检测到0.1-0.5 拷贝/ng RNA水平的融合转录本(图7 ),为血液瘤患者提供更好的辅助诊断、用药及预后提示。目前,该产品已获得国家发明专利授权。
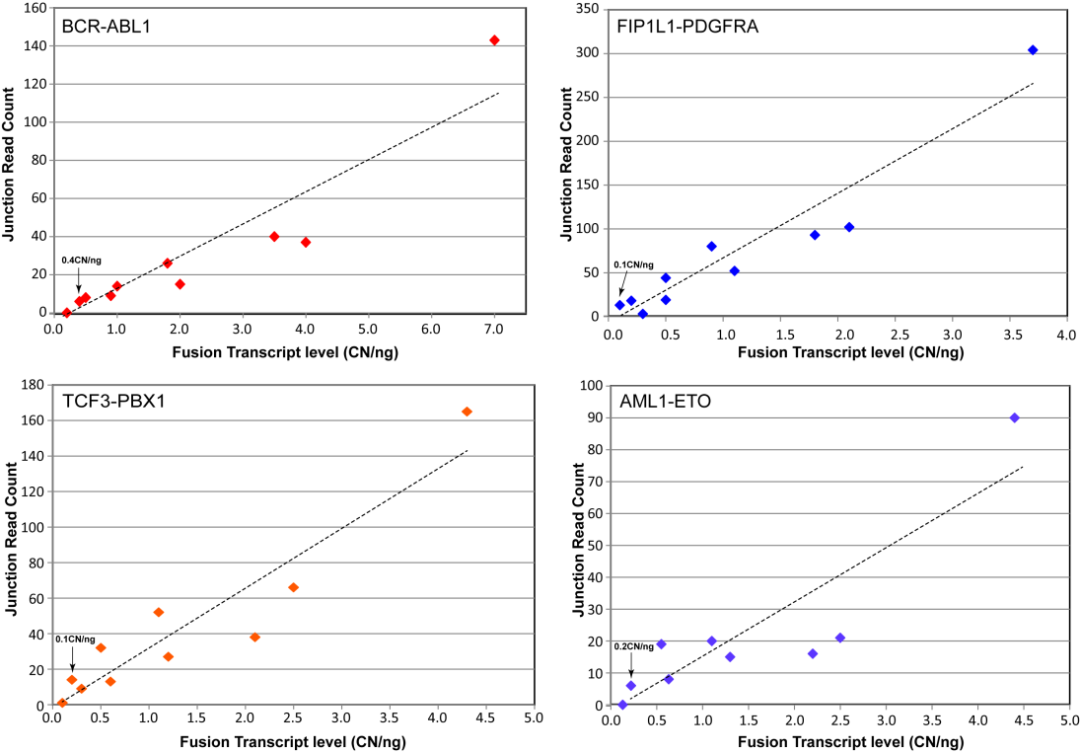
图7 融合转录本的检测性能验证
迈杰转化医学作为国内伴随诊断整体解决方案的领导者,致力于解决精准医疗药物研发及患者用药痛点,围绕生物标志物转化医学研究和伴随诊断开发,建立了完善的核酸组学、蛋白组学、细胞组学技术平台。迈杰转化医学是全球伴随诊断领头创新企业,期待与您的合作!更多详情请致电400-007-1121或MARKETING@MEDxTMC.com。
欢迎关注公司官方网站:
药企合作:www.MEDxTMC.com
产品/检测:www.MEDxTMC.cn
参考文献:
[1] Targetable kinase-activating lesions in Ph-like acute lymphoblastic leukemia.[J]. N Engl J Med, 2014, 371(11):1005-1015.
[2] Tasian S K , Loh M L , Hunger S P . Philadelphia chromosome-like acute lymphoblastic leukemia[J]. Blood, 2017:blood-2017-06-743252.
[3] Seo J S , Ju Y S , Lee W C , et al. The transcriptional landscape and mutational profile of lung adenocarcinoma[J]. Genome Research, 2012, 22(11):2109.
[4] Heyer E E , Deveson I W , Wooi D , et al. Diagnosis of fusion genes using targeted RNA sequencing[J]. Nature Communications, 2019, 10(1).
[5] Mercer T R , Clark M B , Crawford J , et al. Targeted sequencing for gene discovery and quantification using RNA CaptureSeq[J]. Nature Protocols.
央视《匠心》栏目视角看迈杰转化医学